染色质包含的DNA链总长可达2米,它们被包裹在非常狭小的被称为细胞的小室里。为了适应这个微小的空间,染色质丝必须以非常精妙的方式进行折叠,并且不能妨碍关键细胞进程(例如基因表达和复制)。研究染色质的空间折叠形式,能够阐明基因调控、染色体形态变化和基因组稳定性,帮助我们研究与基因组不稳定相关的疾病机制。
目前,有多种技术可用于观察染色质的空间组织,大致可以分为显微观察和分子分析。高级显微镜可以在纳米级下观察染色质的结构,但不能了解该结构中的DNA序列。荧光原位杂交(FISH)可以将染色质结构和DNA序列对应起来,但通量有限,只能同时分析少量的基因位点[1]。基于染色体构象捕获(3C)的分子检测可分析染色质结构和DNA序列,直接检测DNA序列即可获得染色质结构信息[2]。为此,科学家基于NGS技术,全面检测3C产物库,结合下游分析,开发出Hi-C技术[3]。
Hi-C数据分析是得出高分辨率染色质图谱的关键。近日,Cleveland诊所Lerner研究所的胡明教授和加州大学圣地亚哥分校医学院路德维希癌症研究所的任兵教授联合在Nature Methods上发表了一篇名为“SnapHiC: a computational pipeline to identify chromatin loops from single-cell Hi-C data”的研究文章,介绍了可从少量单细胞Hi-C数据中准确地高分辨率识别染色质环结构的SnapHiC软件。
文章发表在Nature Methods
单细胞Hi-C(scHi-C)测序技术被用于单个类型细胞中研究染色质结构,是研究组织中异质性细胞的有力工具。但目前仍缺乏从scHi-C数据中以高分辨率分析染色质环的工具,因此研究团队开发出一种名为SnapHiC的计算方法。这是一种为scHi-C数据定制的计算方法,可从少量单细胞中以高分辨率和高准确度识别染色质环。
SnapHiC通过估算每个细胞中(以10kb每个)基本单位(bin)之间的染色质内接触概率。随后,根据线性基因组距离对估算的接触概率进行归一化。SnapHiC将成对t检验应用于所有细胞的归一化接触概率矩阵,以识别一组细胞中接触概率高于预期的候选“基本单位(bin)”对(或循环候选)。最后,SnapHiC 将“循环候选”分组为集群并确定每个集群内的峰顶。在SnapHiC中,单个细胞被视为独立的数据集,可以估计细胞群内接触频率的可变性,以提高循环检测的统计能力,尤其是当细胞数量较少时。(图1)
图1. SnapHiC的工作流程及分析效果。来源:Nature[4]
研究人员利用来自742个小鼠胚胎干细胞(mES)的scHi-C数据,将 SnapHiC 与Hi-C数据计算工具HiCCUPS进行了基准测试。除了完整的742个细胞外,研究团队还从该数据集中随机抽取了10、25、50、75、100、200、300、400、500、600和700个细胞,对于每个子采样数据集。结果显示,SnapHiC发现的染色质环数量高于HiCCUPS,表明SnapHiC的灵敏度高于HiCCUPS(图1b)。通过F1分数评估方法的整体性能,发现SnapHiC可获得比HiCCUPS更高的F1分数(图1c)。
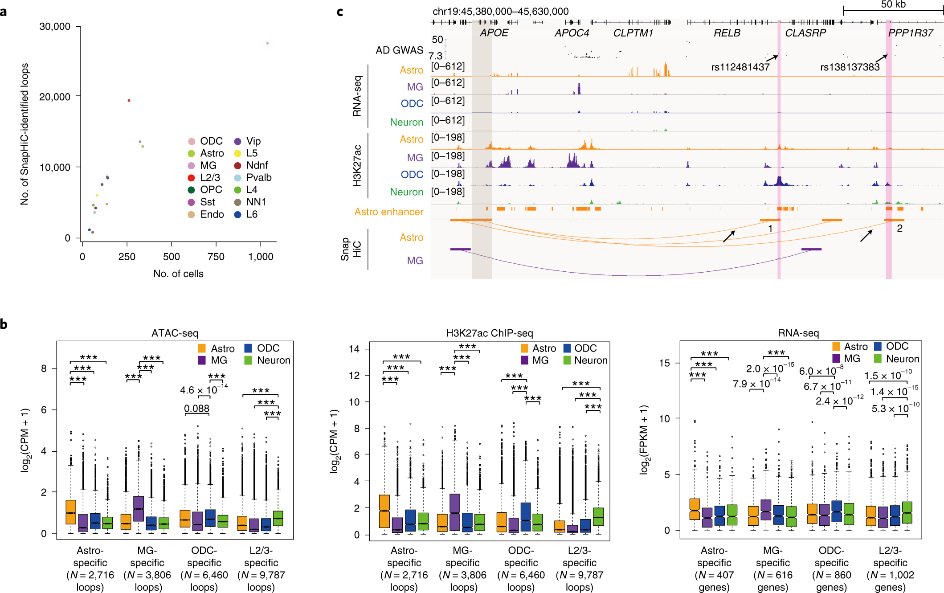
图2.将SnapHiC应用于复杂组织数据,揭示了不同脑细胞类型中的染色质环。来源:Nature[4]
为证明SnapHiC在复杂组织上的使用,研究人员将其应用于已发表的单核甲基-3C-seq(sn-m3C-seq)数据。该数据由人类前额叶脑皮层组织生成,该数据同时分析了来自相同组织的DNA甲基化和染色质。将SnapHiC应用于14个细胞簇中每种细胞簇的Hi-C数据,并以10kb的分辨率识别出大约817-27,379个环(图2a)。研究发现,细胞类型特异性染色质环在匹配的细胞类型中显示出明显高于不匹配细胞类型的信号(图2b )。以基因APOE为示例,该基因在星形胶质细胞中特异性表达。两个星形胶质细胞特异性环将APOE的转录起始位点连接到星形胶质细胞中含有阿尔茨海默病相关SNP的两个活性增强子(rs112481437和rs138137383)。结果表明,APOE可能是这两个SNP的靶基因,特别是在星形胶质细胞中(图2c)。
研究团队设计的SnapHiC算法可以从少量细胞的scHi-C数据集中以高分辨率和高准确度识别染色质环。对来自mES细胞的已发布scHi-C数据的重新分析表明,SnapHiC极大地提高了对染色质环的检测能力。将SnapHiC应用于来自人类前额叶皮层细胞的sn-m3C-seq数据,揭示了细胞类型特异性染色质环,可用于预测非编码SNP的靶基因。SnapHiC有助于促进复杂组织中细胞类型特异性染色质空间组织的研究。
检测大量细胞数据成本高昂,以上研究数据和分析证明了SnapHiC的高灵敏度,且对于复杂组织中发现的稀有细胞类型也具有明显优势。染色质环使DNA 序列中的调控因子能够接触基因并影响其表达,因此能够识别和研究这些染色质环可以增加对相关疾病病理的认识。
参考资料:
[1]. Solovei, I., Cavallo, A., Schermelleh, L., Jaunin, F., Scasselati, C., Cmarko, D., ... & Cremer, T. (2002). Spatial preservation of nuclear chromatin architecture during three-dimensional fluorescence in situ hybridization (3D-FISH). Experimental cell research, 276(1), 10-23.
[2]. Dekker, J., Rippe, K., Dekker, M., & Kleckner, N. (2002). Capturing chromosome conformation. science, 295(5558), 1306-1311.
[3]. Belton, J. M., McCord, R. P., Gibcus, J. H., Naumova, N., Zhan, Y., & Dekker, J. (2012). Hi–C: a comprehensive technique to capture the conformation of genomes. Methods, 58(3), 268-276.
[4]. Yu, M., Abnousi, A., Zhang, Y., Li, G., Lee, L., Chen, Z., ... & Hu, M. (2021). SnapHiC: a computational pipeline to identify chromatin loops from single-cell Hi-C data. Nature Methods, 1-4.